La révolution des CRISPR est en marche
Hélène Gilgenkrantz*
Institut Cochin, Inserm U1016, CNRS UMR 8104, Université Paris-Descartes, 24, rue du faubourg saint-Jacques, 75005 Paris, France
* helene.gilgenkrantz@inserm.fr
Depuis la découverte du ciblage par recombinaison homologue dans les cellules ES (embryonic stem cells), ajouter, enlever, modifier les bases dans une séquence d’ADN était devenu banal, ou presque ! Presque, car il fallait à peu près un an pour obtenir des animaux mutés spécifiquement à l’endroit choisi du génome, en utilisant des constructions complexes transfectées dans les cellules ES. Vinrent ensuite les nucléases à doigts de zinc, les méganucléases (→) [1] et enfin les TALEN dont m/s s’est fait l’écho en début d’année 2014, tant l’approche technologique en semblait novatrice (→) [2]. Mais la révolution des génomes est en marche et l’avènement des CRISPR (clustered regularly interspaced short palindromic repeat) est venu détrôner, en moins de trois ans, toutes les autres approches ! Il suffit, pour s’en convaincre, de dénombrer les articles utilisant cette méthode depuis sa description initiale en tant qu’outil de modification du génome (près de 900 publications). Mais alors, en dehors de leur nom, un peu « crisp……ant » à prononcer, qu’a donc de si révolutionnaire ce nouvel outil du mécano-moléculaire moderne ?
(→) Voir la Nouvelle de M. Cohen-Tannoudji et J.L. Guénet, m/s n° 4, avril 2011, page 387
(→) Voir la Synthèse de B. Dupret et P.O. Angrand, m/s n° 2, février 2014, page 186
Entrée en scène des CRISPR, sortie des nucléases TALE
Pour mieux comprendre le buzz qui agite depuis moins d’un an les journaux, qu’il s’agisse de Science [3–7] ou du Monde1, revenons un peu en arrière et énumérons les caractéristiques requises pour modifier à façon le génome, à la Prévert. Donc, « Pour faire le portrait d’un tel outil, peindre d’abord… » un ciseau moléculaire capable d’induire une cassure double brin de l’ADN en un site choisi du génome. Les deux systèmes de réparation de l’ADN existants, soit par jonction d’extrémités non homologues (NHEJ), soit par recombinaison homologue dirigée (HDR), permettront d’apporter des modifications de l’ADN au site de cassure (insertion, mutation, délétion). Tous les outils précédemment cités assuraient cette fonction de ciseau moléculaire. Pour ne citer que les plus récents en lice, les TALEN (transcription activator-like nuclease) sont produits à partir d’un TALE (transcription activator-like effector) et d’une nucléase, appelée Fokl. Les domaines Fok1 fonctionnent sous forme de dimère, ce qui oblige à obtenir deux constructions avec des domaines de liaison à l’ADN dirigés contre des sites du génome présentant des orientations tête-bêche et un espacement correct [2]. Même si des améliorations techniques ont permis un assemblage plus rapide, la fréquence de la recombinaison reste variable, et surtout la modification de l’ADN dépend du couple ADN-protéine, comme c’est également le cas pour les nucléases à doigts de zinc ou les méganucléases. Or, l’ingénierie des nucléases reste coûteuse et difficile. Voilà la nouveauté extraordinaire des CRISPR : tout en étant extrêmement efficace, le type de reconnaissance dépend d’un couple ADN-ARN : rien de plus facile à fabriquer (Tableau I) !
Analyse comparative de différentes méthodes d’édition du génome.
Mais remontons un peu dans le temps. L’histoire des CRISPR date de la fin des années 1980. Une équipe japonaise découvre alors une séquence d’ADN chez la bactérie E. coli, qui comprend un fragment d’ADN ressemblant à un bactériophage entre deux séquences répétées palindromiques. Ce n’est qu’au milieu des années 2000 que l’on découvre qu’il s’agit là d’un système immunitaire primitif de bactéries leur permettant de se défendre contre les infections virales [3]. En effet, l’ARN transcrit à partir de cette séquence (crARN) subit une maturation en une bibliothèque de petits ARN contenant chacun une séquence guide dérivée du génome de bactériophages virulents. Chacun de ces petits ARN est lié à des protéines associées (Cas) qui sont des nucléases, et les complexes ainsi formés patrouillent dans l’environnement cellulaire à la recherche de l’envahisseur porteur du génome viral homologue. Si la bonne cible est identifiée, elle sera clivée par la nucléase, l’empêchant de poursuivre sa route et protégeant d’autant la cellule de l’infection.
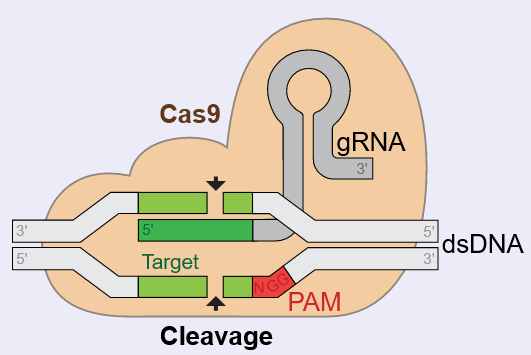
Il faudra attendre encore quelques années pour qu’une chercheuse française, Emmanuelle Charpentier, et son équipe, qui travaillaient alors sur la bactérie Streptococcus Pyogènes, identifient chez celle-ci un système CRISPR dépendant d’une seule nucléase Cas9. Un duplex est formé entre le crARN et un ARN transactivateur, appelé tracrARN, qui active Cas9 et guide l’endonucléase vers la cible [8]. La même équipe montre par la suite que le clivage de chacun des brins de l’ADN requiert ce duplex d’ARN : crARN-guide et tracrARN [4], qui tous deux peuvent être associés en une seule molécule guide. Cas9 coupera la séquence d’ADN cible sur les deux brins seulement si celle-ci est flanquée d’une séquence de trois nucléotides NGG, appelée PAM (proto-spacer adjacent motif), présente à une fréquence élevée dans le génome tant elle est peu discriminante [9] (Figure 1). Tout s’emballe alors rapidement. La première démonstration de l’utilisation du système CRISPR-Cas9 pour modifier spécifiquement le génome de cellules eucaryotes date de 2013 [5, 6].
Principe d’editing par le système CRISPR-Cas9. A. Cassure double brin par le système CRISPR-Cas9. La nucléase Cas 9 (en orange) est guidée par une séquence comprenant une séquence ARN homologue à la séquence cible (en rouge) et une séquence appelée trcrARN (en bleu). La nucléase CRISPR-Cas9 cible une séquence de 3 nucléotides de type NGG appelée PAM (en vert), ouvrant l’ADN complémentaire à la séquence guide (en noir) et créant un hétéroduplex, sgARN-ADN cible. Cas9 comprend deux domaines nucléasiques coupant chacun un des brins d’ADN. B. Construction multiplex. Un seul plasmide peut contenir plusieurs séquences guide (gARN) ainsi que la séquence codant pour Cas9 et un gène de résistance à un antibiotique (ABR).
En résumé, la nucléase Cas9, guidée par un ARN guide (sgARN) qui se lie au locus génomique de choix à proximité d’un motif PAM, crée une cassure double brin qui sera réparée soit par NHEJ, pouvant être à l’origine d’insertion ou de délétion, soit par HDR qui permettra une modification précise du génome au locus en question (Figure 1A).
Les cofondateurs du système d’édition basé sur cette technologie CRISPR-Cas9 ont rapidement rendu les plasmides d’expression accessibles à la communauté scientifique (Addgene.org, Cambridge Massachusetts, dont un recensement récent fait état d’une distribution de cette technologie à plus de 3 000 laboratoires dans le monde). Ainsi, moins de six mois après sa découverte, la modification de lignées cellulaires humaines et murines par ce système a été rapportée avec une efficacité au moins équivalente à celle des TALEN et des nucléases à doits de zinc [5, 6], voire même supérieure [10].
Vous avez dit Révolution ?
Bien sûr, par rapport aux techniques presque ancestrales de transfection de cellules ES réimplantées dans un blastocyste, plus de six mois seront gagnés en modifiant directement le zygote. Cependant, les TALEN étaient également efficaces pour créer des invalidations géniques chez la souris de cette manière. Toutes sortes d’espèces sont modifiables par les CRISPR. Là encore, les TALEN ont aussi été utilisés pour modifier veau, vache, cochon et presque couvée ! Car l’avantage essentiel du système CRISPR-Cas9 sur les autres systèmes utilisant des nucléases ZFN ou les TALEN est ailleurs : il ne dépend pas d’un couple protéine/ADN pour reconnaître la cible, mais d’un couple ARN/ADN, et la construction est devenue un jeu d’enfant (Tableau I). Les constructions peuvent directement être introduites sous forme d’ARN. Par ailleurs, comparativement à toutes les approches utilisant des siARN ou shARN pour éteindre l’expression d’un gène dans un tissu ou un type cellulaire donné, CRISPR est beaucoup plus efficace puisqu’il atteint l’intégralité des cellules descendantes de la cellule cible en ciblant la séquence génomique. Enfin, il est possible de modifier facilement plusieurs cibles simultanément en ajoutant plusieurs complexes guides par une approche dite multiplexing permettant, tel le « vaillant petit tailleur » de Grimm, d’invalider trois gènes d’un coup ou plus dans un même embryon (Figure 1B). De plus, les constructions sont devenues d’une simplicité élémentaire : on pouvait faire l’extraction de l’ADN dans sa cuisine, on aura bientôt le « kitchen-CRISPR » !
Reste la question délicate du risque de clivage dans un site non souhaité, susceptible de modifier un gène non désiré ou de submerger la machinerie de réparation de l’ADN. Cet artéfact est appelé off-target dans le jargon des bricoleurs moléculaires et n’est pas facilement quantifiable quelle que soit l’approche nucléasique utilisée. Or, il a tout d’abord été décrit que plus la concentration de Cas9 était élevée, plus l’efficacité du clivage de la cible était importante, mais plus celui-ci risquait également d’atteindre des cibles non spécifiques. Il semble désormais que les off-targets soient limitées à des sites restreints, homologues au guide sgARN flanqué par une PAM [9].
Des applications en tous genres
L’engouement pour cette approche fut tel que les enjeux financiers sont devenus énormes, plus d’une dizaine d’entreprises de biotechnologie ayant mis sur le marché des produits ou services dérivés des CRISPR, avec toutes les questions complexes de propriété intellectuelle associées à ce type de percée technologique. En attendant de les résoudre, les applications ont déjà fait florès et un nombre considérable d’équipes se sont appropriées cette technique en à peine quelques mois [11]. Voici un florilège d’applications, toutes publiées dans des revues à fort impact factor depuis le début de l’année 2014, à croire que la simple utilisation de l’approche CRISPR-Cas9 donne un blanc-seing pour publier dans le Graal des journaux scientifiques ! Bien sûr, la création de modèles expérimentaux (knock-out et knock-in) vient en tête ; deux petits macaques transgéniques porteurs de deux gènes simultanément inactivés ont ainsi fait le tour du monde [12]. L’injection intraveineuse du cocktail magique ciblant des oncogènes ou des gènes suppresseurs de tumeurs chez la souris a permis d’obtenir des modèles de tumorigenèse hépatique avec une rapidité à faire pâlir tous ceux qui les avaient ciblés précédemment par l’intermédiaire de cellules ES [13]. La réparation de mutations pathogènes suscite naturellement des espoirs thérapeutiques immenses : ainsi, une cataracte génétique de souris induite par une mutation dominante [14] ou des cellules intestinales humaines de patients porteurs d’une mucoviscidose [15] ont-elles été corrigées par cette approche. En fusionnant Cas9 à une protéine fluorescente, il est également possible de suivre in vivo la dynamique des chromosomes en des loci spécifiquement choisis [16]. Enfin, dans le domaine de la génomique à haut débit, la technique CRISPR a permis de cibler de 20 000 à 70 000 gènes susceptibles de conférer une résistance à une chimiothérapie ou à des toxines bactériennes [7, 17]. La possibilité d’introduire facilement des polymorphismes au niveau du génome ou de modifier notre épigénome, en particulier dans un but thérapeutique, devrait théoriquement être réalisable, en ciblant des histones par exemple, permettant également d’entrevoir des pistes de recherche intéressantes.
À l’image de la découverte des enzymes de restriction, également faite par hasard sur des bactéries possédant des endonucléases, il y aura probablement un avant et un après CRISPR. Pourtant, s’il avait fallu parier sur les répercussions majeures d’un mode de défense immunitaire bactérien, il est fort probable qu’aucun comité évaluateur de subvention ANR n’aurait donné son aval.… Une preuve de plus - mais en faut-il encore ? - que la recherche fondamentale d’aujourd’hui nourrit les avancées technologiques de demain. ‡
Liens d’intérêt
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
1
Florence Rosier. Révolution dans l’ingénierie des gènes. Le Monde « Science et techno », 9 juin 2014.
Références
Cohen-Tannoudji M, Guénet JL. Une nouvelle ère pour la génétique du rat. Med Sci (Paris) 2011 ; 27 : 387–390. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
Dupret B, Angrand PO. L’ingénierie des génomes par les TALEN. Med Sci (Paris) 2014 ; 30 : 186–193. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
R. Barrangou. Cas9 targeting and the CRIPSR révolution. Science 2014 ; 344 : 707–708. [CrossRef] [PubMed] [Google Scholar]
Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science 2012 ; 337 : 816–821. [CrossRef] [PubMed] [Google Scholar]
Le Cong, Ran FA, Cox D, et al. Multiplex génome engineering using CRISPR/Cas systems. Science 2013 ; 339 : 819–823. [CrossRef] [PubMed] [Google Scholar]
Mali P, Yang L, Esvelt KM, et al. RNA-guided human génome engineering via Cas9. Science 2013 ; 339 : 823–826. [CrossRef] [PubMed] [Google Scholar]
Shalem O, Sanjana NE, Hartenian E, et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science 2014 ; 343 : 84–87. [CrossRef] [PubMed] [Google Scholar]
Deltcheva E, Chylinski K, Sharma CM, et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature 2011 ; 471 : 602–607. [CrossRef] [PubMed] [Google Scholar]
Sternberg SH, Redding S, Jinek M, Greene EC, Doudna JA. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature 2014 ; 507 : 62–67. [CrossRef] [PubMed] [Google Scholar]
Ding Q, Regan SN, Xia Y, et al. Enhanced efficiency of human pluripotent stem cell genome editing through replacing TALENs with CRISPRs. Cell Stem Cell 2013 ; 12 : 393–394. [CrossRef] [PubMed] [Google Scholar]
Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering. Cell 2014 ; 157 : 12162–12178. [Google Scholar]
Niu Y, Shen B, Cui Y, et al. Generation of gene-modified cynomolgus monkey via Cas9/RNA-mediated gene targeting in one-cell embryos. Cell 2014 ; 156 : 836–843. [CrossRef] [PubMed] [Google Scholar]
Xue W, Chen S, Yin H, et al. CRISPR-mediated direct mutation of cancer genes in the mouse liver. Nature 2014 ; 514 : 380–384. [CrossRef] [PubMed] [Google Scholar]
Wu Y, Liang D, Wang Y, et al. Correction of a genetic disease in a mouse via use of CRISPR-Cas9. Cell Stem Cell 2013 ; 13 : 659–662. [CrossRef] [PubMed] [Google Scholar]
Schwank G, Koo BK, Sasselli V, et al. Functional Repair of CFTR by CRISPR/Cas9. Cell Stem Cell 2013 ; 13 : 653–658. [CrossRef] [PubMed] [Google Scholar]
Chen B, Gilbert LA, Cimini BA, et al. Dynamic imagingof genomic loci inliving human cellz by an optimized CRISPR/Cas system. Cell 2013 ; 155 : 1479–1491. [CrossRef] [PubMed] [Google Scholar]
Koike-Yusa H, Li Y, Tan EP, Velasco-Herrera C, Yusa K. Genome-wide recessive genetic screening in mammalian cells with a lentiviral CRISPR-guide RNA library. Nat Biotechnol 2014 ; 32 : 267–273. [CrossRef] [PubMed] [Google Scholar]